FastKing gDNA Dispelling RT SuperMix
ຄຸນລັກສະນະ
■ໄວ: ຂັ້ນຕອນດຽວເພື່ອສໍາເລັດການກໍາຈັດກໍາມະພັນແລະການຖອດຂໍ້ຄວາມປີ້ນກັບຄືນໄດ້ຢ່າງມີປະສິດທິພາບພາຍໃນ 18 ນາທີໂດຍການເພີ່ມແມ່ແບບເທົ່ານັ້ນ.
efficiency ປະສິດທິພາບສູງ: ການປ່ຽນ transcriptase ແບບປີ້ນກັບກັນຖືກດັດແປງດ້ວຍ motif ແບບ hydrophobic, ມີປະສິດທິພາບ RT ເກີນ 95%.
■ງ່າຍດາຍແລະງ່າຍ: DNase ທີ່ມີຄວາມທົນທານຕໍ່ອຸນຫະພູມສະເພາະມີຜົນກະທົບໄວ, ປະສິດທິພາບສູງດ້ວຍເວລາປະຕິກິລິຍາທີ່ສັ້ນກວ່າ, ແລະຈະບໍ່ມີຜົນກະທົບຕໍ່ກັບ cDNA.
ສະເປັກ
ປະເພດ: Gene ດັດແກ້ transcriptase ຍ້ອນກັບ, gDNase
ຂັ້ນຕອນ: ຂັ້ນຕອນດຽວ (ການກໍາຈັດ DNA ພັນທຸກໍາແລະ RT)
ປະສິດທິພາບ RT: > 95%
ແມ່ແບບ: 1 ng- 2 μg
ເວລາປະຕິບັດງານ: ~ 18 ນທ
ຄໍາຮ້ອງສະຫມັກ: cDNA ທີ່ຖອດຖອນໄດ້ສາມາດໃຊ້ໄດ້ໃນ PCR ທຳ ມະດາ, PCR ເວລາຈິງ, ການກໍ່ສ້າງຫ້ອງສະຸດ cDNA, SAGE (ການວິເຄາະ Serial ຂອງ Gene Expression), ການຂະຫຍາຍເບື້ອງຕົ້ນແລະການທົດລອງ ທຳ ມະດາອື່ນ other.
ຜະລິດຕະພັນທັງຫມົດສາມາດໄດ້ຮັບການປັບແຕ່ງສໍາລັບ ODM/OEM. ສໍາລັບລາຍລະອຽດ,ກະລຸນາຄລິກບໍລິການລູກຄ້າ (ODM/OEM)
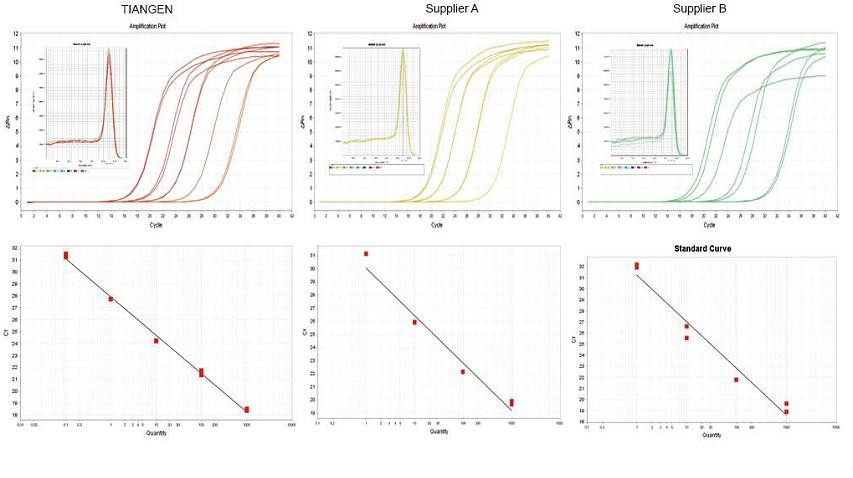 |
ຕົວຢ່າງການທົດລອງ 1. cDNA ໄດ້ຖືກສັງເຄາະໂດຍນໍາໃຊ້ນໍ້າຢາປີ້ນກັບກັນດ້ານປະລິມານ ໜຶ່ງ ຂັ້ນຕອນຂອງ TIANGEN FastKing gDNA Dispelling RT SuperMix, ຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງຈາກຜູ້ຈໍາ ໜ່າຍ A ແລະຜູ້ຈໍາ ໜ່າຍ B ຕາມລໍາດັບ. ກວດພົບເຊື້ອ RN5 ຂອງ ໜູ ທີ່ໃຊ້ TIANGEN Talent qPCR PreMix (SYBR Green), ແລະເສັ້ນໂຄ້ງການຂະຫຍາຍ, ໂຄ້ງການລະລາຍແລະໂຄ້ງມາດຕະຖານໄດ້ຖືກວິເຄາະ. ຜົນໄດ້ຮັບສະແດງໃຫ້ເຫັນວ່າ TIANGEN FastKing gDNA Dispelling RT SuperMix ມີມູນຄ່າ Ct ປະລິມານສູງສຸດຫຼັງຈາກການຖອດຂໍ້ຄວາມກັບຄືນແລະການຕໍ່ຕ້ານຄວາມກົດດັນທີ່ດີເລີດ, ແລະມີຂໍ້ໄດ້ປຽບທີ່ຊັດເຈນສໍາລັບແມ່ແບບທີ່ມີສານຕົກຄ້າງສູງ. |
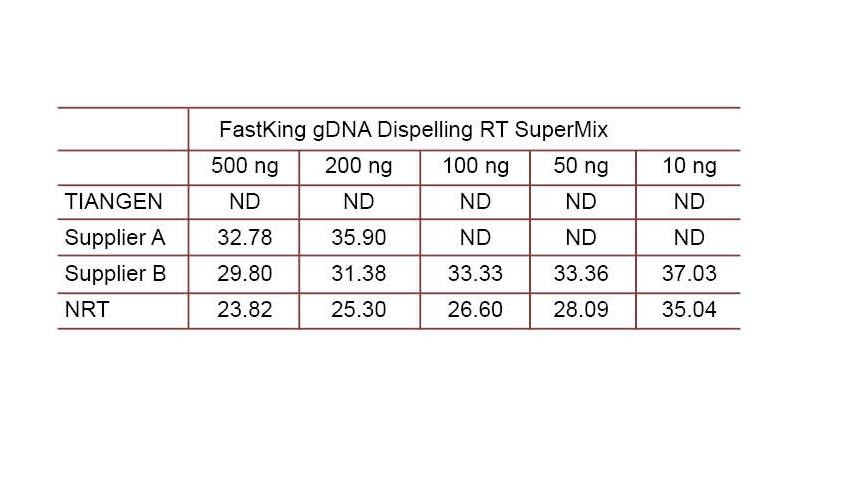 |
ຕົວຢ່າງການທົດລອງ 2. cDNA ໄດ້ຖືກສັງເຄາະໂດຍໃຊ້ນ້ ຳ ຢາປະລິມານແບບປີ້ນກັບກັນເທື່ອລະຂັ້ນຈາກ TIANGEN FastKing gDNA Dispelling RT SuperMix, ຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງຈາກຜູ້ສະ ໜອງ A ແລະຜູ້ສະ ໜອງ B ຕາມ ລຳ ດັບ. ກວດພົບພັນທຸ ກຳ HsG ຂອງມະນຸດໂດຍໃຊ້ TIANGEN Talent qPCR PreMix (SYBR Green), ແລະເພີ່ມຄວາມເຂັ້ມຂົ້ນຂອງ DNA ພັນທຸ ກຳ ທີ່ແຕກຕ່າງກັນດ້ວຍຕົນເອງເພື່ອກວດຫາຄວາມສາມາດໃນການ ກຳ ຈັດ gDNA ຂອງນ້ ຳ ຢາທີ່ແຕກຕ່າງກັນ. ຜົນການຄົ້ນຫາ Ct ສະແດງໃຫ້ເຫັນວ່າ TIANGEN FastKing gDNA Dispelling RT SuperMix ມີຄວາມສາມາດດີເລີດໃນການກໍາຈັດ DNA ພັນທຸກໍາ. ເຖິງ 500 ng ຂອງສິ່ງເສດເຫຼືອ DNA ພັນທຸ ກຳ ສາມາດຖືກ ກຳ ຈັດອອກໄດ້ຢ່າງສົມບູນໂດຍບໍ່ມີຜົນກະທົບຕໍ່ຜົນໄດ້ຮັບ. ND: ບໍ່ກວດພົບ. NRT: ການກວດຫາສ່ວນປະສົມໂດຍບໍ່ມີການຖອດຂໍ້ຄວາມປີ້ນກັບກັນ. |
A-1 RNA ເສື່ອມສະພາບ
—— ເຮັດຄວາມສະອາດ RNA ຄຸນນະພາບສູງໂດຍບໍ່ມີການປົນເປື້ອນ. ວັດສະດຸທີ່ສານສະກັດຈາກ RNA ຄວນຈະສົດເທົ່າທີ່ຈະເປັນໄປໄດ້ເພື່ອປ້ອງກັນການເສື່ອມສະພາບຂອງ RNA. ວິເຄາະຄວາມສົມບູນຂອງ RNA ຢູ່ໃນເຈລທີ່ບໍ່ມີຕົວຕົນກ່ອນປະຕິກິລິຍາ RT. ຫຼັງຈາກການສະກັດເອົາ RNA, ມັນຄວນເກັບຮັກສາໄວ້ໃນຮູບແບບ 100%. ຖ້າ RNase inhibitor ຖືກນໍາໃຊ້, ອຸນຫະພູມຄວາມຮ້ອນຄວນຈະ <45 ° C, ແລະ pH ຄວນຈະຕ່ໍາກວ່າ 8.0, ຖ້າບໍ່ດັ່ງນັ້ນຕົວຍັບຍັ້ງຈະປ່ອຍ RNase ທີ່ຜູກມັດໄວ້ທັງົດ. ຍິ່ງໄປກວ່ານັ້ນ, RNase inhibitor ຄວນຖືກເພີ່ມເຂົ້າໃນວິທີແກ້ໄຂບັນຈຸມີ≥ 0.8 mM DTT.
A-2 RNA ມີສານຍັບຍັ້ງການປະຕິກິລິຍາການຖອດຂໍ້ຄວາມກັບຄືນ
- —Reverse transcription inhibitors ປະກອບມີ SDS, EDTA, glycerol, sodium pyrophosphate, spermidine, formamide, guanidine salt, ແລະອື່ນ Mix. ລ້າງນ້ ຳ Rົນ RNA ດ້ວຍເອທານອນ 70% (v/v) ເພື່ອ ກຳ ຈັດສານຍັບຍັ້ງ.
A-3 ການຫຼໍ່ຫຼອມທາດໄຄແຜ່ນທີ່ບໍ່ພຽງພໍທີ່ໃຊ້ສໍາລັບການສັງເຄາະສາຍພັນທໍາອິດຂອງ cDNA
—— ກຳ ນົດວ່າອຸນຫະພູມໃນການຫຼໍ່ແມ່ນເsuitableາະສົມກັບເຄື່ອງປູພື້ນທີ່ໃຊ້ໃນການທົດລອງ. ສຳ ລັບ hexamers ແບບສຸ່ມ, ແນະ ນຳ ໃຫ້ຮັກສາອຸນຫະພູມຢູ່ທີ່ 25 ° C ເປັນເວລາ 10 ນາທີກ່ອນທີ່ຈະເຖິງອຸນຫະພູມປະຕິກິລິຍາ. ສໍາລັບຕົວກໍານົດສະເພາະຂອງພັນທຸກໍາ (GSP), ລອງໃຊ້ GSP ອື່ນ, ຫຼືສະຫຼັບໄປເປັນ oligo (dT) ຫຼື hexamer ແບບສຸ່ມ.
A-4 ຈໍານວນນ້ອຍ of ຂອງການເລີ່ມ RNA
—— ເພີ່ມປະລິມານຂອງ RNA. ສໍາລັບຕົວຢ່າງ RNA ໜ້ອຍ ກວ່າ 50 ng, 0.1 μgຫາ 0.5 μg acetyl BSA ສາມາດຖືກນໍາໃຊ້ໃນການສັງເຄາະ cDNA ສາຍທໍາອິດ.
A-5 ລໍາດັບເປົ້າisາຍບໍ່ໄດ້ຖືກສະແດງອອກໃນເນື້ອເຍື່ອທີ່ວິເຄາະ.
—— ລອງໃຊ້ແພຈຸລັງອື່ນ.
ປະຕິກິລິຍາ PCR A-6 ລົ້ມເຫລວ
—— ສໍາລັບ RT-PCR ສອງຂັ້ນຕອນ, ແມ່ແບບ cDNA ໃນຂັ້ນຕອນ PCR ບໍ່ສາມາດເກີນ 1/5 ຂອງປະລິມານການໂຕ້ຕອບ.
A-1 ການຫລໍ່ຫລອມແບບທີ່ບໍ່ສະເພາະເຈາະຈົງຂອງທາດແລະແມ່ແບບ
—— ແຜ່ນຮອງໄ' 3'-end ບໍ່ຄວນມີ 2-3 dG ຫຼື dC. ໃຊ້ primers ສະເພາະ Gene ໃນການສັງເຄາະສາຍທໍາອິດແທນການໃຊ້ primers ແບບສຸ່ມຫຼື oligo (dT). ໃຊ້ອຸນຫະພູມໃນການຫຼໍ່ສູງຂຶ້ນໃນສອງສາມຮອບທໍາອິດ, ແລະຈາກນັ້ນອຸນຫະພູມໃນການຫຼໍ່ຫຼອມຕໍ່າກວ່າ. ໃຊ້ໂປຣແກມ polymerase Taq DNA ເລີ່ມຕົ້ນທີ່ຮ້ອນສໍາລັບ PCR ເພື່ອປັບປຸງສະເພາະຂອງປະຕິກິລິຍາ.
A-2 ການອອກແບບທີ່ບໍ່ດີຂອງຕົວສະເພາະເຈາະຈົງ
—— ປະຕິບັດຕາມຫຼັກການດຽວກັນສໍາລັບການອອກແບບ primer ຂະຫຍາຍ.
A-3 RNA ປົນເປື້ອນດ້ວຍ DNA ພັນທຸ ກຳ
—— ປິ່ນປົວ RNA ດ້ວຍ PCR-DNase I. ຈັດຕັ້ງປະຕິກິລິຍາຄວບຄຸມໂດຍບໍ່ມີການຖອດຂໍ້ຄວາມກັບຄືນເພື່ອກວດພົບການປົນເປື້ອນ DNA.
A-4 ການປະກອບຂອງ dimer primer
—— ການອອກແບບ primers ໂດຍບໍ່ມີການຈັດລຽງລໍາດັບເສີມຢູ່ທ້າຍ 3 '.
A-5 Mg ສູງເກີນໄປ2+ ຄວາມເຂັ້ມຂົ້ນ
—— ເພີ່ມປະສິດທິພາບ Mg2+ ຄວາມເຂັ້ມຂົ້ນຂອງແຕ່ລະແມ່ແບບແລະການປະສົມປະສານເບື້ອງຕົ້ນ
A-6 ປົນເປື້ອນດ້ວຍ DNA ຕ່າງປະເທດ
—— ໃຊ້ຄໍາແນະນໍາທີ່ທົນທານຕໍ່ອາກາດລະລາຍແລະເອນໄຊ UDG.
A-1 ເນື້ອໃນຂອງຜະລິດຕະພັນຂອງເຊືອກທໍາອິດແມ່ນສູງເກີນໄປ
—— ຫຼຸດປະລິມານຂອງຜະລິດຕະພັນສາຍພັນທໍາອິດໃນຂັ້ນຕອນປະຕິກິລິຍາ PCR ແບບດັ້ງເດີມ.
A-2 ຈໍານວນ primer ສູງເກີນໄປໃນປະຕິກິລິຍາ PCR
—— ຫຼຸດການປ້ອນຂໍ້ມູນເບື້ອງຕົ້ນລົງ.
A-3 ຮອບວຽນຫຼາຍເກີນໄປ
—— ເພີ່ມປະສິດທິພາບເງື່ອນໄຂປະຕິກິລິຍາ PCR ແລະຫຼຸດຈໍານວນຮອບວຽນ PCR.
A-4 ອຸນຫະພູມໃນການຫຼໍ່ຫຼອມຕໍ່າເກີນໄປ
—— ເພີ່ມອຸນຫະພູມໃນການຫຼໍ່ຫຼອມເພື່ອປ້ອງກັນການເລີ່ມຕົ້ນແລະການຂະຫຍາຍທີ່ບໍ່ສະເພາະເຈາະຈົງ.
A-5 ການຂະຫຍາຍທີ່ບໍ່ແມ່ນສະເພາະຂອງຊິ້ນສ່ວນ oligonucleotide ທີ່ສ້າງຂຶ້ນໂດຍການເສື່ອມສະພາບຂອງ DNase ຂອງ DNA-ສະກັດເອົາ RNA ທີ່ມີຄຸນນະພາບສູງເພື່ອປ້ອງກັນການປົນເປື້ອນຂອງ DNA.
RT-PCR ແມ່ນເພື່ອປີ້ນຄືນການຖ່າຍທອດ RNA ເຂົ້າໄປໃນ cDNA, ແລະຫຼັງຈາກນັ້ນໃຊ້ cDNA ທີ່ຖອດຖອນໄດ້ດ້ານຫຼັງເປັນແມ່ແບບສໍາລັບປະຕິກິລິຍາ PCR ເພື່ອຂະຫຍາຍຊິ້ນສ່ວນເປົ້າາຍ. ເລືອກເອົາ primers ແບບສຸ່ມ, Oligo dT ແລະ primers ສະເພາະຂອງ gene ອີງຕາມເງື່ອນໄຂສະເພາະຂອງການທົດລອງ. ຕົວຊ່ວຍເບື້ອງຕົ້ນທັງaboveົດຂ້າງເທິງນີ້ສາມາດຖືກນໍາໃຊ້ສໍາລັບ mRNA ຈຸລັງ eukaryotic ສັ້ນໂດຍບໍ່ມີໂຄງສ້າງຂອງ hairpin.
primer ແບບສຸ່ມ: ເforາະສົມກັບ RNA ຍາວທີ່ມີໂຄງສ້າງຂອງ hairpin, ພ້ອມທັງ RNA ທຸກປະເພດເຊັ່ນ: rRNA, mRNA, tRNA, ແລະອື່ນ They.
Oligo dT: ເforາະສົມກັບ RNA ທີ່ມີ PolyA tailing (prokaryotic RNA, eukaryotic Oligo dT rRNA ແລະ tRNA ບໍ່ມີຫາງ PolyA). ເນື່ອງຈາກວ່າ Oligo dT ຖືກຜູກມັດກັບຫາງ PolyA, ຄຸນນະພາບຂອງຕົວຢ່າງ RNA ແມ່ນຕ້ອງການສູງ, ແລະແມ້ແຕ່ການເສື່ອມສະພາບເລັກນ້ອຍກໍ່ຈະຫຼຸດປະລິມານການສັງເຄາະ cDNA ເຕັມຄວາມຍາວໄດ້ຢ່າງຫຼວງຫຼາຍ.
primer ສະເພາະເຈາະຈົງ: ສົມທົບກັບລໍາດັບແມ່ແບບ, ເsuitableາະສົມກັບສະຖານະການທີ່ຮູ້ລໍາດັບເປົ້າາຍ.
ມີສອງວິທີ:
1. ວິທີການອ້າງອີງພາຍໃນ: ໃນທາງທິດສະດີ, cDNA ແມ່ນ DNA fragments ທີ່ມີຄວາມຍາວແຕກຕ່າງກັນ, ສະນັ້ນຜົນຂອງ electrophoresis ແມ່ນ smear. ຖ້າຄວາມອຸດົມສົມບູນຂອງ RNA ຕໍ່າ, ບໍ່ມີຜະລິດຕະພັນໃດຈະສະແດງອອກໃນ electrophoresis, ແຕ່ນີ້ບໍ່ໄດ້meanາຍຄວາມວ່າບໍ່ມີຜະລິດຕະພັນໃດຈະຖືກຂະຫຍາຍອອກໂດຍ PCR. ໂດຍທົ່ວໄປ, ການອ້າງອີງພາຍໃນສາມາດຖືກນໍາໃຊ້ເພື່ອກວດຫາ cDNA. ຖ້າການອ້າງອີງພາຍໃນມີຜົນໄດ້ຮັບ, ຄຸນະພາບຂອງ cDNA ສາມາດໄດ້ຮັບການຮັບປະກັນໂດຍພື້ນຖານ (ໃນບາງກໍລະນີ, ຖ້າຊິ້ນສ່ວນພັນທຸກໍາເປົ້າisາຍຍາວເກີນໄປ, ອາດຈະມີຂໍ້ຍົກເວັ້ນ).
2. ຖ້າມີການຮູ້ຈັກຂະຫຍາຍພັນທຸ ກຳ ໂດຍແມ່ແບບນີ້, ມັນສາມາດກວດພິສູດໄດ້ໂດຍຕົວຊ່ວຍຂອງເຊື້ອສາຍນີ້. ການຂະຫຍາຍການອ້າງອີງພາຍໃນບໍ່ໄດ້necessarilyາຍຄວາມວ່າບໍ່ມີບັນຫາກັບ cDNA. ເນື່ອງຈາກວ່າການອ້າງອີງພາຍໃນມີຄວາມອຸດົມສົມບູນສູງໃນ cDNA, ມັນງ່າຍທີ່ຈະຂະຫຍາຍ. ຖ້າ cDNA ຖືກເຊື່ອມໂຊມບາງສ່ວນດ້ວຍເຫດຜົນຕ່າງ various, ຈາກທັດສະນະຂອງຄວາມເປັນໄປໄດ້, ຜົນໄດ້ຮັບ PCR ຂອງພັນທຸກໍາເປົ້າabundາຍຄວາມອຸດົມສົມບູນຕໍ່າຈະໄດ້ຮັບຜົນກະທົບຢ່າງຫຼວງຫຼາຍ. ໃນຂະນະທີ່ການອ້າງອີງພາຍໃນຍັງມີຄວາມອຸດົມສົມບູນສູງ, ການຂະຫຍາຍສຽງອາດຈະບໍ່ໄດ້ຮັບຜົນກະທົບ.
ເສື່ອມສະພາບບາງສ່ວນຂອງ RNA. ກວດຫາຄວາມສົມບູນແລະເຮັດໃຫ້ RNA ບໍລິສຸດ
ເນື້ອໃນ RNA ຂອງຊະນິດພັນທີ່ແຕກຕ່າງກັນອາດຈະແຕກຕ່າງກັນ, ແຕ່ໂດຍທົ່ວໄປແລ້ວ, RNA ທັງextractົດທີ່ສະກັດມາຄວນມີສອງແຖບ 28S ແລະ 18S ທີ່ຊັດເຈນໃນ electrophoresis gel, ແລະຄວາມສະຫວ່າງຂອງແຖບກ່ອນ ໜ້າ ນີ້ຄວນຈະສູງກວ່າສອງເທົ່າໃນຍຸກສຸດທ້າຍ. ແຖບ 5S ຊີ້ໃຫ້ເຫັນວ່າ RNA ໄດ້ເຊື່ອມໂຊມລົງ, ແລະຄວາມສະຫວ່າງຂອງມັນແມ່ນອັດຕາສ່ວນກັບລະດັບຂອງການເຊື່ອມໂຊມ. ການຂະຫຍາຍການອ້າງອີງພາຍໃນທີ່ປະສົບຜົນ ສຳ ເລັດບໍ່ໄດ້meanາຍຄວາມວ່າບໍ່ມີບັນຫາກັບ RNA, ເພາະວ່າການອ້າງອີງພາຍໃນແມ່ນມີຄວາມອຸດົມສົມບູນສູງ, RNA ສາມາດຂະຫຍາຍອອກໄດ້ຕາບໃດທີ່ການເຊື່ອມໂຊມບໍ່ຮ້າຍແຮງ. OD ໄດ້260/ອຈ280ອັດຕາສ່ວນຂອງ RNA ບໍລິສຸດທີ່ວັດແທກດ້ວຍ spectrophotometer ຄວນຢູ່ລະຫວ່າງ 1.9 ຫາ 2.1. ປະລິມານທາດໂປຼຕີນທີ່ບໍ່ສະອາດໃນ RNA ໜ້ອຍ ໜຶ່ງ ຈະເຮັດໃຫ້ອັດຕາສ່ວນຫຼຸດລົງ. ຕາບໃດທີ່ຄ່າບໍ່ຕໍ່າເກີນໄປ, RT ຈະບໍ່ໄດ້ຮັບຜົນກະທົບ. ສິ່ງທີ່ ສຳ ຄັນທີ່ສຸດ ສຳ ລັບ RT ແມ່ນຄວາມສົມບູນຂອງ RNA.
ການຂະຫຍາຍພັນທຸ ກຳ ອ້າງອີງພາຍໃນສາມາດຊີ້ບອກໄດ້ວ່າ RT ໄດ້ປະສົບຜົນ ສຳ ເລັດ, ແຕ່ມັນບໍ່ ຈຳ ເປັນຕ້ອງກ່ຽວຂ້ອງກັບຄຸນນະພາບຂອງສາຍພັນ cDNA. ເນື່ອງຈາກວ່າສ່ວນທີ່ອ້າງອີງພາຍໃນໂດຍທົ່ວໄປມີຂະ ໜາດ ນ້ອຍແລະສູງໃນການສະແດງອອກ, ພວກມັນງ່າຍກວ່າທີ່ຈະປະສົບຜົນສໍາເລັດໃນການຖອດຂໍ້ຄວາມຖອຍຫຼັງ. ແນວໃດກໍ່ຕາມ, ຂະ ໜາດ ແລະການສະແດງອອກຂອງພັນທຸ ກຳ ເປົ້າvarາຍແຕກຕ່າງກັນຈາກ ກຳ ມະພັນຫາພັນທຸ ກຳ. ຄຸນະພາບ cDNA ບໍ່ສາມາດຖືກຕັດສິນໄດ້ໂດຍການອ້າງອີງພາຍໃນໂດຍສະເພາະສໍາລັບຊິ້ນສ່ວນເປົ້າlongerາຍທີ່ຍາວກວ່າ 2 kb.
ຕົວຢ່າງບາງອັນມີໂຄງສ້າງມັດທະຍົມທີ່ຊັບຊ້ອນ, ຫຼືມີເນື້ອໃນ GC ອຸດົມສົມບູນ, ຫຼືມີຄ່າທີ່ມີຄວາມອຸດົມສົມບູນຕໍ່າ. ໃນກໍລະນີເຫຼົ່ານີ້, ຄວນຄັດເລືອກ transcriptase ທີ່ເreverseາະສົມຕາມຂະ ໜາດ ຂອງຊິ້ນສ່ວນເປົ້າandາຍແລະຕົວຢ່າງ. ສໍາລັບແມ່ແບບ RNA ທີ່ມີເນື້ອໃນ GC ສູງແລະໂຄງສ້າງມັດທະຍົມທີ່ຊັບຊ້ອນ, ມັນເປັນການຍາກທີ່ຈະເປີດໂຄງສ້າງຂັ້ນສອງຢູ່ໃນອຸນຫະພູມຕໍ່າ, ຫຼືມີ transcriptase ຍ້ອນກັບທົ່ວໄປ. ສໍາລັບແມ່ແບບເຫຼົ່ານີ້, Quant Reverse Transcriptase ສາມາດເລືອກໄດ້, ເນື່ອງຈາກວ່າປະສິດທິພາບການຖອດຂໍ້ຄວາມຍ້ອນກັບຂອງມັນແມ່ນເຫັນໄດ້ຊັດເຈນດີກວ່າຊຸດ M-MLV series transcriptase ປີ້ນກັບກັນ, ເຊິ່ງສາມາດປີ້ນຄືນການຖອດຂໍ້ຄວາມແມ່ແບບ RNA ຕ່າງ efficient ໄດ້ຢ່າງມີປະສິດທິພາບແລະຖ່າຍທອດ RNA ເຂົ້າໄປໃນ cDNA ສາຍທໍາອິດໃນຂອບເຂດສູງສຸດ. ເມື່ອນໍາໃຊ້ຊຸດ transcriptase ປີ້ນກັບຄືນທົ່ວໄປ, ລະບົບ 20 μlສາມາດປະຕິເສດການຖອດຂໍ້ຄວາມໄດ້ຢ່າງ ໜ້ອຍ 1 μgຂອງ RNA ທັງົດ. ກະລຸນາເອົາໃຈໃສ່ກັບຄວາມສາມາດ RT ສູງສຸດຂອງຊຸດ. ຖ້າແມ່ແບບຖືກເພີ່ມເຂົ້າໄປຫຼາຍເກີນໄປ, ການຖອດຂໍ້ຄວາມປີ້ນກັບກັນຈະເປັນທີ່ນິຍົມໃຫ້ກັບ RNA ທີ່ມີຄວາມອຸດົມສົມບູນສູງ. ດັ່ງນັ້ນ, ມັນດີກວ່າບໍ່ໃຫ້ເກີນຄວາມຈຸສູງສຸດຂອງລະບົບ.
A-1 ກໍານົດວ່າ RNA ຖືກເຊື່ອມໂຊມລົງຢ່າງຮຸນແຮງແລະຖ້າ RT ປະສົບຜົນສໍາເລັດ
ໂດຍທົ່ວໄປ, ເຫດຜົນສໍາລັບຄວາມລົ້ມເຫຼວຂອງການຂະຫຍາຍການອ້າງອີງພາຍໃນແມ່ນມັກເກີດຈາກການເຊື່ອມໂຊມຂອງ RNA ຮ້າຍແຮງ. ອີກເຫດຜົນ ໜຶ່ງ ທີ່ເປັນໄປໄດ້ແມ່ນການຖອດຂໍ້ຄວາມລົ້ມເຫລວ. ການອ້າງອີງພາຍໃນບໍ່ສາມາດຖືກນໍາໃຊ້ເປັນມາດຕະຖານເພື່ອຕັດສິນຄຸນນະພາບຂອງສາຍເຊ ລຳ ດຽວຂອງ cDNA, ແຕ່ມັນສາມາດຖືກນໍາໃຊ້ເປັນມາດຕະຖານເພື່ອຕັດສິນວ່າການຖອດຂໍ້ຄວາມກັບຄືນໄດ້ສໍາເລັດຫຼືບໍ່ຖ້າບໍ່ມີບັນຫາກ່ຽວກັບຄຸນນະພາບຂອງ RNA. ສິ່ງທີ່ສໍາຄັນທີ່ສຸດໃນຂະບວນການຖອດຂໍ້ຄວາມປີ້ນກັບກັນແມ່ນການຮັກສາອຸນຫະພູມຄົງທີ່ແລະລະບົບປະຕິກິລິຍາຄົງທີ່ເພື່ອປັບປຸງປະສິດທິພາບຂອງປະຕິກິລິຍາ.
A-2 ຕັດສິນກໍານົດວ່າ primers ສໍາລັບການຂະຫຍາຍພັນທຸກໍາກະສານອ້າງອີງພາຍໃນແມ່ນເຊື່ອຖືໄດ້ຫຼືບໍ່ແລະຖ້າມີບັນຫາຫຍັງກັບນໍ້າຢາທີ່ໃຊ້ຢູ່ໃນ PCR.
ສໍາລັບປະລິມານທີ່ກ່ຽວຂ້ອງ, RNA ຕ້ອງໄດ້ຮັບການປະລິມານກ່ອນການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ, ເຊິ່ງຈໍາເປັນຕ້ອງມີຢູ່ໃນຫຼາຍຊຸດການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ, ຍົກຕົວຢ່າງ, ຄໍານວນປະລິມານການປ້ອນເຂົ້າ RNA ເປັນ 1 μg. ເນື່ອງຈາກວ່າ cDNA ທີ່ຖອດຖອນໄດ້ແມ່ນເປັນການແກ້ໄຂແບບປະສົມ, ລວມທັງ RNA, oligo dT, enzyme, dNTP, ແລະແມ້ກະທັ້ງ DNA ຕົກຄ້າງ ໜ້ອຍ ໜຶ່ງ, ຄວາມບ່ຽງເບນຈະເກີດຂຶ້ນ, ສະນັ້ນມັນເປັນໄປບໍ່ໄດ້ທີ່ຈະກໍານົດປະລິມານ cDNA ໄດ້ຢ່າງຖືກຕ້ອງ. ສະນັ້ນ, ປະລິມານ RNA ແມ່ນມີຄວາມ ຈຳ ເປັນ. ພິຈາລະນາປະສິດທິພາບການຖອດຂໍ້ມູນແບບປີ້ນກັບກັນແມ່ນຄືກັນລະຫວ່າງຕົວຢ່າງທີ່ແຕກຕ່າງກັນ, ປະລິມານຂອງ cDNA ທີ່ໄດ້ຮັບຄວນຈະຄືກັນ, ແລະການວິເຄາະດ້ານປະລິມານສາມາດສະແດງໃຫ້ເຫັນການປຽບທຽບລະດັບການສະແດງອອກຂອງພັນທຸກໍາທີ່ແຕກຕ່າງກັນໃນຈໍານວນທັງRົດຂອງ RNA ທັງົດ. ເມື່ອປະຕິບັດ PCR ດ້ານປະລິມານ fluorescence, cDNA ດ້ານປະລິມານອາດຈະບໍ່ຈໍາເປັນຫຼັງຈາກການຖອດຂໍ້ຄວາມຍ້ອນກັບເພາະວ່າເຊື້ອສາຍອ້າງອີງພາຍໃນສາມາດເຮັດ ໜ້າ ທີ່ອ້າງອີງໄດ້.
ມັນສ່ວນໃຫຍ່ກ່ຽວຂ້ອງກັບພັນທຸ ກຳ, ແລະການຖອດຂໍ້ຄວາມຂອງຊິ້ນສ່ວນຍາວ reverse ແມ່ນບໍ່ເປັນໄປໄດ້ ສຳ ລັບພັນທຸ ກຳ ສ່ວນໃຫຍ່. ປະການທໍາອິດ, ປະສິດທິພາບຂອງການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນແມ່ນຕໍ່າກວ່າ PCR. ອັນທີສອງ, ພາກພື້ນທີ່ອຸດົມສົມບູນຂອງ GC ແລະໂຄງສ້າງຂັ້ນສອງຂອງພັນທຸກໍາຈໍາກັດທັງການຖ່າຍທອດຄືນແລະ PCR. ສຸດທ້າຍ, ຄວາມຊື່ສັດແລະປະສິດທິພາບການຂະຫຍາຍຂອງ PCR ແມ່ນຍາກທີ່ຈະຮັບປະກັນໄດ້ໃນເວລາດຽວກັນ. ໃນຂະບວນການຖອດຂໍ້ຄວາມກັບຄືນ, ບໍ່ມີໃຜສາມາດຮັບປະກັນໄດ້ວ່າຈະໄດ້ຊິ້ນທີ່ຍາວນານສໍາລັບພັນທຸກໍາສໍາເນົາທີ່ຕໍ່າ, ໂດຍສະເພາະການໃຊ້ oligo dT. ສຳ ລັບ UTR 5 'ທີ່ມີ GC ຫຼາຍ, ມັນຍິ່ງຍາກກວ່າ. ສະນັ້ນ, ມັນຍັງເປັນວິທີການທີ່ສົມເຫດສົມຜົນທີ່ຈະປີ້ນຄືນຂໍ້ມູນຈາກການຂຽນເບື້ອງຕົ້ນແບບສຸ່ມ, ຊອກຫາສະຖານທີ່ທໍາລາຍທໍາມະຊາດຢູ່ໃນຊິ້ນສ່ວນເປົ້າ,າຍ, ຂະຫຍາຍອອກເປັນແຕ່ລະສ່ວນ, ແລະຈາກນັ້ນດໍາເນີນການຈໍາກັດການຍ່ອຍອາຫານແລະການລອກ. ໂດຍທົ່ວໄປ, ມັນເປັນການຍາກທີ່ຈະຂະຫຍາຍຊິ້ນສ່ວນທີ່ໃຫຍ່ກວ່າ 2 kb ໂດຍກົງ, ແຕ່ມັນບໍ່ເປັນໄປບໍ່ໄດ້ສະເtoີໄປທີ່ຈະໄດ້ຮັບ: 1. ກ່ອນອື່ນ,ົດ, ຮັບປະກັນຄວາມສົມບູນຂອງ RNA/mRNA, ແລະການສະກັດເອົາ TRIZOL ແມ່ນມັກ. ຊຸດ 2.M-MLV RT-PCR ສາມາດຖືກນໍາໃຊ້ໂດຍກົງ. ຂະຫຍາຍເວລາການຫລອມແລະເພີ່ມຈໍານວນຮອບວຽນໃນຂະບວນການຂະຫຍາຍຢ່າງຖືກຕ້ອງ. ອີກທາງເລືອກ ໜຶ່ງ, PCR ທີ່ຕິດຢູ່ສາມາດ ນຳ ໃຊ້ໄດ້, ຫຼືປະຕິບັດ ໜຶ່ງ ຫຼືສອງປະຕິກິລິຍາກ່ອນດ້ວຍການຂະຫຍາຍເວລາແລະການຂະຫຍາຍເວລາທີ່ເbeforeາະສົມກ່ອນການຂະຫຍາຍ PCR ປົກກະຕິ, ເຊິ່ງອາດຈະຊ່ວຍຂະຫຍາຍຊິ້ນສ່ວນຕ່າງ. ເອົາໃຈໃສ່ກັບຄວາມຊື່ສັດຂອງ polymerase. 3. Long Taq ສາມາດໃຊ້ໃນ PCR ເພື່ອໃຫ້ໄດ້ຜົນທີ່ເidealາະສົມ. 4. ສໍາລັບຄໍາຮ້ອງສະຫມັກການສະແດງອອກທາດໂປຼຕີນ, polymerase ຄວາມຊື່ສັດສູງຄວນໄດ້ຮັບການນໍາໃຊ້.
ມີສອງປະເພດຂອງ transcriptase ປີ້ນກັບກັນທີ່ສະ ເໜີ ໂດຍ TIANGEN: Quant/King RTase ແລະ TIANScript M-MLV. ຄວາມແຕກຕ່າງຕົ້ນຕໍລະຫວ່າງເຂົາເຈົ້າແມ່ນ ຈຳ ນວນແມ່ແບບປ້ອນເຂົ້າ. Quant ແມ່ນເອກະລັກ transcriptase ຍ້ອນກັບ, ເຊິ່ງແຕກຕ່າງຈາກ M-MLV ທີ່ໃຊ້ທົ່ວໄປມາຈາກເຊື້ອໄວຣັສ leukemia Moloney murine. Quant ແມ່ນປະສິດທິພາບສູງ transcriptase ປີ້ນກັບຄືນມາໃefficiency່ທີ່ມີປະສິດທິພາບສູງສະແດງໃຫ້ເຫັນເປັນປະ ຈຳ ໂດຍວິສະວະ ກຳ Escherichia coli. ປະລິມານແມ່ນເsuitableາະສົມ ສຳ ລັບການຂະຫຍາຍ 50 ng-2 μgຂອງ RNA ດ້ວຍກິດຈະ ກຳ ການຖອດຂໍ້ຄວາມກັບຄືນໄດ້ສູງແລະໃຫ້ຜົນຜະລິດສູງ. ເມື່ອປຽບທຽບກັບ MMLV ຫຼື AMV ທຳ ມະດາ, ລັກສະນະໃຫຍ່ທີ່ສຸດຂອງ Quant ແມ່ນວ່າມັນມີຄວາມ ສຳ ພັນທີ່ເຂັ້ມແຂງຫຼາຍກັບແມ່ແບບ RNA ແລະສາມາດປີ້ນຄືນແມ່ແບບທີ່ສັບສົນໂດຍບໍ່ມີການປ່ຽນແປງຂອງອຸນຫະພູມສູງ. ສໍາລັບແມ່ແບບທີ່ມີເນື້ອໃນ GC ສູງກວ່າ, ປະສິດທິພາບການປີ້ນກັບກັນແມ່ນສູງກວ່າ. ແນວໃດກໍ່ຕາມ, transcriptase ປີ້ນກັບກັນນີ້ມີກິດຈະກໍາ RNase H, ເຊິ່ງອາດຈະກະທົບກັບຄວາມຍາວຂອງຜະລິດຕະພັນ cDNA (ເsuitableາະສົມສໍາລັບແມ່ແບບ <4.5 kb). ສຳ ລັບການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນແບບ ທຳ ມະດາ, ແນະ ນຳ ໃຫ້ໃຊ້ TIANScript MMLV reverse transcriptase. RTase ນີ້ແມ່ນເອນໄຊທີ່ຖືກດັດແກ້ທີ່ມີກິດຈະກໍາ RNase H ທີ່ອ່ອນແອຫຼາຍ, ເຊິ່ງເsuitableາະສົມກັບການສັງເຄາະ cDNA ຍາວ (> 5 kb).
ການຖອດຂໍ້ຄວາມປີ້ນກັບກັນຂັ້ນຕອນດຽວແລະການຂະຫຍາຍ PCR ແມ່ນສໍາເລັດໃນທໍ່ດຽວກັນໂດຍບໍ່ເປີດcoverາປົກທໍ່ລະຫວ່າງການສັງເຄາະ cDNA ແລະການຂະຫຍາຍ, ເຊິ່ງເປັນປະໂຫຍດເພື່ອຫຼຸດການປົນເປື້ອນ. ເນື່ອງຈາກຕົວຢ່າງ cDNA ທັງobtainedົດທີ່ໄດ້ມາແມ່ນໃຊ້ເພື່ອຂະຫຍາຍ, ຄວາມອ່ອນໄຫວແມ່ນສູງກວ່າ, ໂດຍມີຕໍາ່ສຸດທີ່ 0.01 pg ຂອງ RNA ທັງົດ. ສໍາລັບ RTPCR ຂັ້ນຕອນ ໜຶ່ງ ທີ່ປະສົບຜົນສໍາເລັດ, ໂດຍທົ່ວໄປແລ້ວ primers ສະເພາະເຈາະຈົງຖືກນໍາໃຊ້ໂດຍທົ່ວໄປເພື່ອລິເລີ່ມການສັງເຄາະ cDNA. ວິທີການສອງຂັ້ນຕອນ, ຄືການຖອດຂໍ້ຄວາມກັບຄືນແລະການຂະຫຍາຍ PCR ແມ່ນດໍາເນີນຢູ່ໃນສອງຂັ້ນຕອນ. ທຳ ອິດການຖອດຂໍ້ຄວາມປີ້ນກັບກັນແມ່ນປະຕິບັດຈາກແມ່ແບບ RNA ເພື່ອໃຫ້ໄດ້ cDNA, ແລະ cDNA ທີ່ໄດ້ຮັບແມ່ນຕ້ອງມີປະຕິກິລິຍາ PCR ທີ່ແຕກຕ່າງກັນຢ່າງ ໜຶ່ງ ຫຼືຫຼາຍອັນ. ວິທີການສອງຂັ້ນຕອນສາມາດນໍາໃຊ້ oligo (dT) ຫຼື primers ແບບສຸ່ມເພື່ອນໍາພາການສັງເຄາະສາຍພັນທໍາອິດຂອງ cDNA, ແລະສາມາດປີ້ນຄືນການຖ່າຍທອດຂໍ້ມູນ mRNA ທັງfromົດຈາກຕົວຢ່າງສະເພາະ.
ປະເພດຜະລິດຕະພັນ
ເປັນຫຍັງເລືອກພວກເຮົາ
ນັບຕັ້ງແຕ່ການສ້າງຕັ້ງ, ໂຮງງານຂອງພວກເຮົາໄດ້ພັດທະນາຜະລິດຕະພັນລະດັບໂລກອັນທໍາອິດໂດຍຍຶດັ້ນຫຼັກການ
ຂອງຄຸນນະພາບຄັ້ງທໍາອິດ. ຜະລິດຕະພັນຂອງພວກເຮົາໄດ້ຮັບຊື່ສຽງໂດ່ງດັງໃນອຸດສາຫະກໍາແລະເປັນທີ່ເຊື່ອຖືໄດ້ໃນບັນດາລູກຄ້າໃand່ແລະເກົ່າ.