ຊຸດ RNA-Seq ທີ່ຄ້າງຢູ່ TIANSeq (ຄວາມສະຫວ່າງ)
ຄຸນລັກສະນະ
uniform ຄວາມເປັນເອກະພາບການຈັດລໍາດັບດີ: ຄວາມຊື່ສັດສູງຂອງການຂະຫຍາຍ PCR ແລະບໍ່ມີອະຄະຕິພື້ນຖານ.
efficiency ປະສິດທິພາບການປ່ຽນຫ້ອງສະ■ຸດສູງ: ການກໍ່ສ້າງຫ້ອງສະefficiencyຸດທີ່ມີປະສິດທິພາບສູງສາມາດຮັບປະກັນໄດ້ສໍາລັບຕົວຢ່າງ 1ng mRNA.
operation ປະຕິບັດງານໄດ້ໄວ: ຂະບວນການກໍ່ສ້າງຫ້ອງສະwholeຸດທັງneedsົດຕ້ອງການພຽງແຕ່ 5.5 ຊົ່ວໂມງເທົ່ານັ້ນ.
ສະເປັກ
ປະເພດ: ການກະກຽມຫ້ອງສະRຸດ RNA ທິດທາງຂອງ NGS
ຕົວຢ່າງ: RNA ທັງົດ
ເປົ້າຫມາຍ: mRNA, lncRNA
ເລີ່ມການປ້ອນຕົວຢ່າງ: ຕົວຢ່າງ RNA ທັງisົດແມ່ນ 10 ng-1 μg, ແລະຕົວຢ່າງຂອງ mRNA ແມ່ນຕໍ່າຄື 1 ng
ເວລາປະຕິບັດງານ: 5.5-6.5 ຊົ່ວໂມງ
ແອັບພລິເຄຊັນທາງລຸ່ມ: ການຈັດ ລຳ ດັບຢູ່ເທິງເວທີ illumina.
ຜະລິດຕະພັນທັງຫມົດສາມາດໄດ້ຮັບການປັບແຕ່ງສໍາລັບ ODM/OEM. ສໍາລັບລາຍລະອຽດ,ກະລຸນາຄລິກບໍລິການລູກຄ້າ (ODM/OEM)
ການປຽບທຽບຜົນຜະລິດຂອງຫ້ອງສະຸດ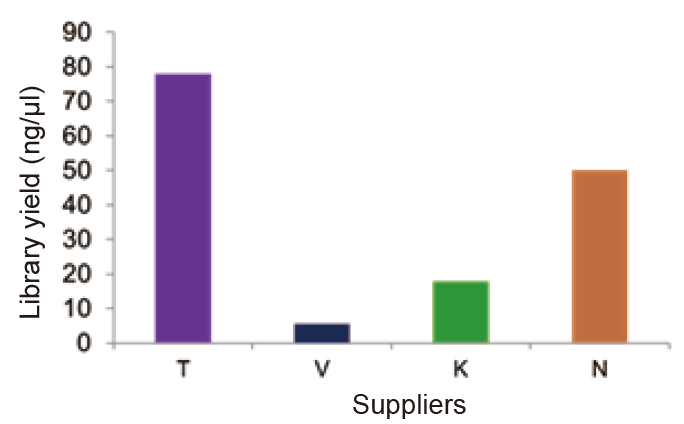 |
ຮູບ 1. ຫ້ອງສະRຸດ RNA ຂອງ 1 μgມະນຸດ 293T ເຊນທັງRົດ RNA ຖືກສ້າງຂຶ້ນໂດຍໃຊ້ TIANSeq Stranded RNASeq Kit ແລະຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງຈາກຜູ້ສະ ໜອງ V, K ແລະ N. , K ແລະ N. |
ການຄຸ້ມຄອງຂໍ້ມູນເອກະພາບ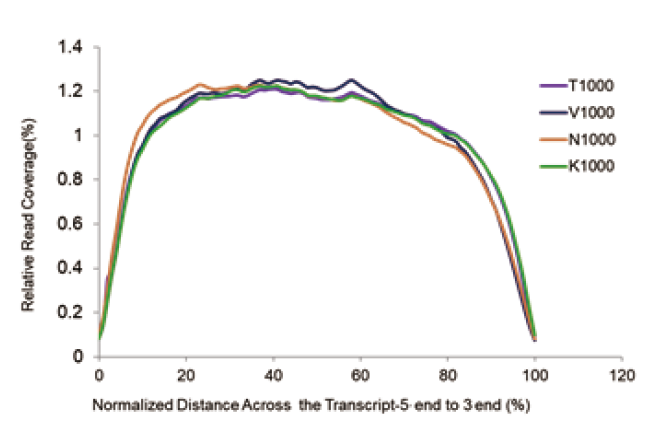 |
ຮູບ 2. ຫ້ອງສະRຸດ RNA ຂອງ 1 μgມະນຸດ 293T ເຊນທັງRົດ RNA ຖືກສ້າງຂຶ້ນໂດຍໃຊ້ TIANSeq Stranded RNASeq Kit ແລະຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງຈາກຜູ້ສະ ໜອງ V, K ແລະ N. ສຸດທ້າຍ, ເຊິ່ງດີກວ່າຜູ້ສະ ໜອງ V, ແລະປະສິດທິພາບແມ່ນຄ້າຍຄືກັນກັບຜະລິດຕະພັນຂອງຜູ້ສະ ໜອງ N ແລະ K. |
ຂອບເຂດຄໍາຮ້ອງສະຫມັກກ້ວາງຂອງວັດສະດຸປ້ອນຕົວຢ່າງ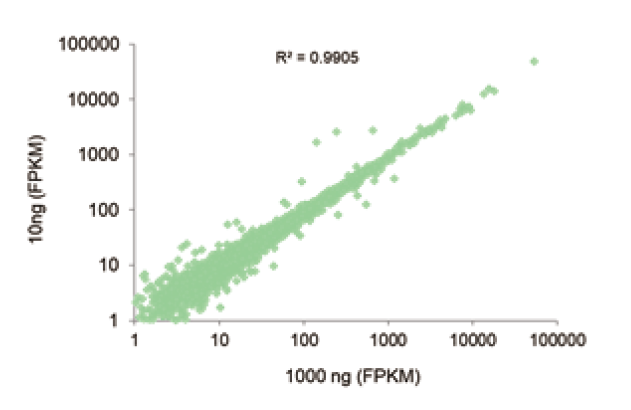 |
ຮູບ 3. ຊຸດ TIANSeq Stranded RNA-Seq ເຂົ້າກັນໄດ້ກັບການປ້ອນເຂົ້າ RNA ທັງ10ົດ 10 ng-1 μg, ແລະຫ້ອງສະconstructedຸດທີ່ສ້າງຂຶ້ນພາຍໃຕ້ຕົວຢ່າງວັດສະດຸຕົວຢ່າງທີ່ແຕກຕ່າງກັນມີຄວາມສອດຄ່ອງສູງ. ຜົນໄດ້ຮັບສະແດງໃຫ້ເຫັນວ່າຕົວຄູນຄວາມສໍາພັນຂອງການສະແດງອອກຂອງເຊື້ອລະຫວ່າງຫ້ອງສະreachedຸດບັນລຸໄດ້ສູງກວ່າ 0.99 ພາຍໃຕ້ການປ້ອນ RNA ທັງofົດຂອງ 10 ng ແລະ 1 μgຂອງ 293T cell, ແລະຄວາມສອດຄ່ອງຂອງຂໍ້ມູນແມ່ນສູງ. |
ປັດຈຸບັນ, ເຕັກໂນໂລຍີການຈັດລໍາດັບຄວາມໄວສູງສ່ວນໃຫຍ່ແມ່ນອີງໃສ່ເທັກໂນໂລຍີການຮຽງລໍາດັບລຸ້ນຕໍ່ໄປ. ເນື່ອງຈາກວ່າຄວາມຍາວການອ່ານຂອງເທັກໂນໂລຍີການຮຽງລໍາດັບລຸ້ນຕໍ່ໄປແມ່ນມີຈໍາກັດ, ພວກເຮົາຕ້ອງແຍກລໍາດັບຄວາມຍາວທັງintoົດອອກເປັນຫ້ອງສະfragຸດຊິ້ນສ່ວນນ້ອຍ to ເພື່ອລໍາດັບ. ອີງຕາມຄວາມຕ້ອງການຂອງການທົດລອງການຈັດລໍາດັບທີ່ແຕກຕ່າງກັນ, ໂດຍປົກກະຕິແລ້ວພວກເຮົາເລືອກການຈັດລໍາດັບຊັ້ນດຽວຫຼືສິ້ນສຸດສອງເທື່ອ. ປະຈຸບັນຊິ້ນສ່ວນ DNA ຂອງຫ້ອງສະsequຸດຈັດ ລຳ ດັບລຸ້ນຕໍ່ໄປໂດຍທົ່ວໄປແມ່ນແຈກຢາຍຢູ່ໃນລະດັບ 200-800 bp.

ກ) DNA ບໍ່ມີຄຸນນະພາບແລະມີສານຍັບຍັ້ງ. ໃຊ້ຕົວຢ່າງ DNA ທີ່ມີຄຸນນະພາບສູງເພື່ອຫຼີກເວັ້ນການສະກັດກັ້ນການເຄື່ອນໄຫວຂອງເອນໄຊ.
ຂ) ຈໍານວນຕົວຢ່າງ DNA ແມ່ນບໍ່ພຽງພໍເມື່ອນໍາໃຊ້ວິທີການທີ່ບໍ່ມີ PCR ເພື່ອສ້າງຫ້ອງສະDNAຸດ DNA. ເມື່ອການປ້ອນຂໍ້ມູນ DNA ທີ່ກະແຈກກະຈາຍເກີນ 50 ng, ຂະບວນການເຮັດວຽກທີ່ບໍ່ມີ PCR ສາມາດເລືອກໄດ້ໃນລະຫວ່າງຂັ້ນຕອນການກໍ່ສ້າງຫ້ອງສະຸດ. ຖ້າຈໍານວນສໍາເນົາຂອງຫ້ອງສະຸດຕໍ່າເກີນໄປທີ່ຈະຈັດລໍາດັບໄດ້ໂດຍກົງ, ຫ້ອງສະDNAຸດ DNA ສາມາດຂະຫຍາຍອອກໄດ້ໂດຍ PCR ຫຼັງຈາກການຕິດຕໍ່ອະແດັບເຕີ.
c) ການປົນເປື້ອນ RNA ນໍາໄປສູ່ການກວດສອບປະລິມານ DNA ເບື້ອງຕົ້ນທີ່ບໍ່ຖືກຕ້ອງການປົນເປື້ອນ RNA ອາດຈະມີຢູ່ໃນຂັ້ນຕອນການກັ່ນຕອງ DNA ພັນທຸກໍາ, ເຊິ່ງອາດຈະເຮັດໃຫ້ປະລິມານ DNA ບໍ່ຖືກຕ້ອງແລະການໂຫຼດ DNA ບໍ່ພຽງພໍໃນລະຫວ່າງການສ້າງຫ້ອງສະຸດ. RNA ສາມາດເອົາອອກໄດ້ໂດຍການປິ່ນປົວດ້ວຍ RNase.
A-1
ກ) ຊິ້ນສ່ວນຂະ ໜາດ ນ້ອຍ (60 bp-120 bp) ປະກົດວ່າຊິ້ນສ່ວນຂະ ໜາດ ນ້ອຍໂດຍປົກກະຕິແລ້ວເປັນຊິ້ນສ່ວນຂອງອະແດັບເຕີຫຼື dimers ທີ່ສ້າງຂຶ້ນໂດຍຕົວແປງ. ການເຮັດໃຫ້ບໍລິສຸດດ້ວຍລູກປັດສະນະແມ່ເຫຼັກຂອງ Agencourt AMPure XP ສາມາດເອົາຊິ້ນສ່ວນເຄື່ອງປັບເຫຼົ່ານີ້ອອກໄດ້ຢ່າງມີປະສິດທິພາບແລະຮັບປະກັນຄຸນນະພາບການຈັດລໍາດັບ.
ຂ. ຖ້າຊິ້ນສ່ວນ DNA ເພີ່ມຂຶ້ນຫຼາຍກ່ວາ 120 bp ຫຼັງຈາກການຕໍ່ສາຍອະແດບເຕີ, ມັນອາດຈະເກີດຈາກການຂະຫຍາຍຊິ້ນສ່ວນຜິດປົກກະຕິຂອງການຂະຫຍາຍ PCR ຫຼາຍເກີນໄປ. ການຫຼຸດຜ່ອນຈໍານວນຂອງຮອບວຽນ PCR ສາມາດປ້ອງກັນສະຖານະການໄດ້.
c) ຂະ ໜາດ ຜິດປົກກະຕິຂອງຊິ້ນສ່ວນ DNA ຂອງຫ້ອງສະafterຸດຫຼັງຈາກການຕໍ່ສາຍໄຟຄວາມຍາວຂອງຕົວດັດແປງໃນຊຸດນີ້ແມ່ນ 60 bp. ເມື່ອສອງສົ້ນຂອງຊິ້ນສ່ວນຖືກເຊື່ອມເຂົ້າກັບອະແດັບເຕີ, ຄວາມຍາວຈະເພີ່ມຂຶ້ນພຽງແຕ່ 120 bp. ເມື່ອໃຊ້ອະແດັບເຕີອື່ນທີ່ບໍ່ໄດ້ສະ ໜອງ ໃຫ້ໂດຍຊຸດນີ້, ກະລຸນາຕິດຕໍ່ຫາຜູ້ສະ ໜອງ ເພື່ອໃຫ້ຂໍ້ມູນທີ່ກ່ຽວຂ້ອງເຊັ່ນ: ຄວາມຍາວຂອງອະແດັບເຕີ. ກະລຸນາຮັບປະກັນວ່າຂັ້ນຕອນການເຮັດວຽກແລະການດໍາເນີນການທົດລອງປະຕິບັດຕາມຂັ້ນຕອນທີ່ໄດ້ອະທິບາຍໄວ້ຢູ່ໃນຄູ່ມື.
d) ຂະ ໜາດ ຊິ້ນສ່ວນ DNA ຜິດປົກກະຕິກ່ອນການຕໍ່ສາຍສາກເຫດຜົນຂອງບັນຫານີ້ອາດເກີດຈາກສະພາບການປະຕິກິລິຍາຜິດພາດໃນລະຫວ່າງການກະຈາຍ DNA. ເວລາຕິກິຣິຍາທີ່ແຕກຕ່າງກັນຄວນຖືກນໍາໃຊ້ສໍາລັບການປ້ອນເຂົ້າ DNA ທີ່ແຕກຕ່າງກັນ. ຖ້າການປ້ອນ DNA ຫຼາຍກ່ວາ 10 ng, ພວກເຮົາແນະນໍາໃຫ້ເລືອກເວລາປະຕິກິລິຍາຂອງ 12 ນາທີເປັນເວລາເລີ່ມຕົ້ນສໍາລັບການເພີ່ມປະສິດທິພາບ, ແລະຂະ ໜາດ ຊິ້ນສ່ວນທີ່ຜະລິດອອກມາໃນເວລານີ້ສ່ວນໃຫຍ່ແມ່ນຢູ່ໃນຊ່ວງ 300-500 bp. ຜູ້ໃຊ້ສາມາດເພີ່ມຫຼືຫຼຸດຄວາມຍາວຂອງຊິ້ນ DNA ເປັນເວລາ 2-4 ນາທີຕາມຄວາມຕ້ອງການຂອງເຂົາເຈົ້າເອງເພື່ອເພີ່ມປະສິດທິພາບຂອງຊິ້ນ DNA ດ້ວຍຂະ ໜາດ ທີ່ຕ້ອງການ.
ກ -2
ກ) ເວລາການກະຈາຍບໍ່ໄດ້ຖືກປັບໃຫ້ເIfາະສົມຖ້າ DNA ທີ່ກະແຈກກະຈາຍມີຂະ ໜາດ ນ້ອຍເກີນໄປຫຼືໃຫຍ່ເກີນໄປ, ກະລຸນາອ້າງອີງເຖິງຂໍ້ແນະນໍາສໍາລັບການເລືອກເວລາການແບ່ງສ່ວນທີ່ໃຫ້ໄວ້ໃນຄໍາແນະນໍາເພື່ອກໍານົດເວລາຕິກິຣິຍາ, ແລະໃຊ້ຈຸດເວລານີ້ເປັນຕົວຄວບຄຸມ, ນອກຈາກນັ້ນຕັ້ງຄ່າ ລະບົບປະຕິກິລິຍາທີ່ຈະຍືດຍາວຫຼືຫຼຸດອອກ 3 ນາທີເພື່ອເຮັດໃຫ້ມີການປັບຕົວທີ່ຖືກຕ້ອງຫຼາຍຂຶ້ນກ່ຽວກັບເວລາການກະຈາຍ.
ກ -3
ການກະຈາຍຂະ ໜາດ ຜິດປົກກະຕິຂອງ DNA ຫຼັງຈາກການປິ່ນປົວດ້ວຍການແບ່ງຕົວ
ກ. ລະລາຍ 5 × Fragmentation Enzyme Mix ນ້ ຳ ຢາກ້ອນ. ເມື່ອລະລາຍແລ້ວ, ໃຫ້ປະສົມນໍ້າຢາໃສ່ສະເlyີກັນໂດຍການຖອດລຸ່ມຂອງທໍ່ຄ່ອຍ gently. ບໍ່ vortex reagent ໄດ້!
b) ຕົວຢ່າງການປ້ອນ DNA ປະກອບດ້ວຍ EDTA ຫຼືມົນລະພິດອື່ນ De ການເຮັດໃຫ້ທາດໄອອອນເກືອandົດແລະຕົວແທນ chelating ໃນຂັ້ນຕອນການກັ່ນຕອງ DNA ແມ່ນມີຄວາມສໍາຄັນເປັນພິເສດຕໍ່ກັບຜົນສໍາເລັດຂອງການທົດລອງ. ຖ້າ DNA ຖືກລະລາຍໃນ 1 × TE, ໃຊ້ວິທີການທີ່ໃຫ້ໄວ້ໃນຄໍາແນະນໍາເພື່ອປະຕິບັດການແຍກຕົວ. ຖ້າຄວາມເຂັ້ມຂົ້ນຂອງ EDTA ຢູ່ໃນການແກ້ໄຂແມ່ນບໍ່ແນ່ນອນ, ຂໍແນະນໍາໃຫ້ເຮັດຄວາມສະອາດ DNA ແລະເຮັດໃຫ້ລະລາຍໃນນໍ້າ deionized ສໍາລັບປະຕິກິລິຍາຕໍ່ມາ.
c) ປະລິມານ DNA ເບື້ອງຕົ້ນທີ່ບໍ່ຖືກຕ້ອງຂະ ໜາດ ຂອງ DNA ທີ່ກະແຈກກະຈາຍແມ່ນກ່ຽວຂ້ອງຢ່າງໃກ້ຊິດກັບປະລິມານການປ້ອນເຂົ້າ DNA. ກ່ອນການປິ່ນປົວການກະແຈກກະຈາຍ, ປະລິມານທີ່ຖືກຕ້ອງຂອງ DNA ໂດຍໃຊ້ Qubit, Picogreen ແລະວິທີການອື່ນ other ແມ່ນເປັນສິ່ງຈໍາເປັນເພື່ອກໍານົດຈໍານວນທີ່ແນ່ນອນຂອງ DNA ໃນລະບົບປະຕິກິລິຍາ.
d) ການກະກຽມລະບົບປະຕິກິລິຍາບໍ່ໄດ້ປະຕິບັດຕາມຄໍາແນະນໍາການກະກຽມລະບົບປະຕິກິລິຍາທີ່ກະແຈກກະຈາຍຕ້ອງດໍາເນີນຢູ່ເທິງນໍ້າກ້ອນຢ່າງເຂັ້ມງວດຕາມຄໍາແນະນໍາ. ເພື່ອຮັບປະກັນຜົນກະທົບທີ່ດີທີ່ສຸດ, ສ່ວນປະກອບຂອງປະຕິກິລິຍາທັງshouldົດຄວນວາງໃສ່ເທິງນໍ້າກ້ອນແລະການກະກຽມລະບົບປະຕິກິລິຍາຄວນດໍາເນີນຫຼັງຈາກຄວາມເຢັນສົມບູນ. ຫຼັງຈາກການກະກຽມສໍາເລັດແລ້ວ, ກະລຸນາຕີຫຼື pipet ປະສົມຢ່າງລະອຽດ. ບໍ່ vortex!
1. ວິທີການປະສົມທີ່ບໍ່ຖືກຕ້ອງ (vortex, ການສັ່ນສະເທືອນທີ່ຮຸນແຮງ, ແລະອື່ນ)) ຈະເຮັດໃຫ້ເກີດການກະຈາຍຜິດປົກກະຕິຂອງຊິ້ນສ່ວນຂອງຫ້ອງສະຸດ (ດັ່ງທີ່ສະແດງຢູ່ໃນຮູບຕໍ່ໄປນີ້), ດັ່ງນັ້ນຈຶ່ງສົ່ງຜົນກະທົບຕໍ່ຄຸນນະພາບຂອງຫ້ອງສະຸດ. ສະນັ້ນ, ເວລາກະກຽມວິທີແກ້ໄຂປະສົມ Fragmentation Mix, ກະລຸນາຄ່ອຍ pip ປີ້ນຂຶ້ນແລະລົງເພື່ອປະສົມ, ຫຼືໃຊ້ປາຍນິ້ວມືປັດແລະປະສົມໃຫ້ເທົ່າກັນ. ຈົ່ງລະມັດລະວັງບໍ່ໃຫ້ປະສົມກັບ vortex.

2. DNA ທີ່ມີຄວາມບໍລິສຸດສູງຕ້ອງຖືກ ນຳ ໃຊ້ເຂົ້າໃນການກໍ່ສ້າງຫ້ອງສະຸດ
integrity ຄວາມສົມບູນຂອງ DNA ດີ: ແຖບ electrophoresis ມີຫຼາຍກວ່າ 30 kb, ໂດຍບໍ່ມີການຫາງ
■ OD260/230:> 1.5
■ OD260/280: 1.7-1.9
3. ປະລິມານການປ້ອນ DNA ຕ້ອງຖືກຕ້ອງມັນໄດ້ຖືກແນະນໍາໃຫ້ນໍາໃຊ້ວິທີການ Qubit ແລະ PicoGreen ເພື່ອກໍານົດປະລິມານ DNA, ແທນທີ່ຈະແມ່ນ Nanodrop.
4. ເນື້ອໃນຂອງ EDTA ຢູ່ໃນວິທີແກ້ໄຂ DNA ຕ້ອງຖືກກໍານົດວ່າ EDTA ມີອິດທິພົນອັນໃຫຍ່ຫຼວງຕໍ່ກັບປະຕິກິລິຍາການແບ່ງສ່ວນ. ຖ້າເນື້ອໃນຂອງ EDTA ສູງ, ການກວດ DNA ຕ້ອງເຮັດໃຫ້ສະອາດກ່ອນການກວດຄັ້ງຕໍ່ໄປ.
5. ວິທີແກ້ໄຂປະຕິກິລິຍາການກະຈາຍຄວາມຮ້ອນຕ້ອງໄດ້ກະກຽມໄວ້ເທິງນ້ ຳ ກ້ອນຂະບວນການກະຈາຍຄວາມອ່ອນໄຫວຕໍ່ກັບອຸນຫະພູມແລະເວລາປະຕິກິລິຍາ (ໂດຍສະເພາະຫຼັງຈາກເພີ່ມເຄື່ອງເພີ່ມເຂົ້າໄປ). ເພື່ອຮັບປະກັນຄວາມຖືກຕ້ອງຂອງເວລາປະຕິກິລິຍາ, ກະລຸນາກຽມລະບົບປະຕິກິລິຍາໃສ່ນໍ້າກ້ອນ.
6. ເວລາປະຕິກິລິຍາຂອງການແບ່ງສ່ວນຈະຕ້ອງຖືກຕ້ອງເວລາປະຕິກິລິຍາຂອງຂັ້ນຕອນການແບ່ງສ່ວນຈະມີຜົນກະທົບໂດຍກົງຕໍ່ຂະ ໜາດ ຂອງຜະລິດຕະພັນທີ່ເປັນຊິ້ນສ່ວນ, ສະນັ້ນສົ່ງຜົນກະທົບຕໍ່ການກະຈາຍຂະ ໜາດ ຂອງຊິ້ນສ່ວນ DNA ໃນຫ້ອງສະຸດ.
1. ຕົວຢ່າງປະເພດໃດທີ່ສາມາດໃຊ້ໄດ້ກັບຊຸດນີ້?
ປະເພດຕົວຢ່າງທີ່ໃຊ້ໄດ້ຂອງຊຸດນີ້ສາມາດເປັນ RNA ທັງorົດຫຼື mRNA ທີ່ບໍລິສຸດດ້ວຍຄວາມສົມບູນຂອງ RNA ດີ. ຖ້າ RNA ທັງisົດຖືກໃຊ້ເພື່ອສ້າງຫ້ອງສະຸດ, ແນະນໍາໃຫ້ໃຊ້ຊຸດການສູນເສຍ rRNA (Cat#4992363/4992364/4992391) ເພື່ອເອົາ rRNA ອອກກ່ອນ.
2. ຕົວຢ່າງ FFPE ສາມາດນໍາໃຊ້ເພື່ອສ້າງຫ້ອງສະwithຸດດ້ວຍຊຸດນີ້ໄດ້ບໍ?
mRNA ໃນຕົວຢ່າງ FFPE ຈະຖືກເຊື່ອມໂຊມລົງໃນລະດັບໃດ ໜຶ່ງ, ດ້ວຍຄວາມສົມບູນທີ່ບໍ່ດີ. ເມື່ອໃຊ້ຊຸດນີ້ສໍາລັບການກໍ່ສ້າງຫ້ອງສະຸດ, ແນະນໍາໃຫ້ໃຊ້ເວລາການກະຈາຍໃຫ້ດີທີ່ສຸດ (ຫຍໍ້ເວລາການກະຈາຍຫຼືບໍ່ປະຕິບັດການແບ່ງສ່ວນ).
3. ການນໍາໃຊ້ຂັ້ນຕອນການຄັດເລືອກຂະ ໜາດ ທີ່ໃຫ້ໄວ້ຢູ່ໃນຄູ່ມືຜະລິດຕະພັນ, ອັນໃດທີ່ອາດຈະເຮັດໃຫ້ພາກສ່ວນທີ່ໃສ່ແລ້ວປະກົດວ່າມີການບ່ຽງເບນເລັກນ້ອຍ?
ການຄັດເລືອກຂະ ໜາດ ຈະຕ້ອງດໍາເນີນຢ່າງເຂັ້ມງວດຕາມຂັ້ນຕອນການເລືອກຂະ ໜາດ ຢູ່ໃນຄູ່ມືຜະລິດຕະພັນນີ້. ຖ້າມີການບ່ຽງເບນ, ເຫດຜົນອາດຈະແມ່ນວ່າລູກປັດສະນະແມ່ເຫຼັກບໍ່ສົມດຸນກັບອຸນຫະພູມຫ້ອງຫຼືບໍ່ຖືກປະສົມເຂົ້າກັນຢ່າງເຕັມທີ່, ທໍ່ສົ່ງທໍ່ບໍ່ຖືກຕ້ອງຫຼືແຫຼວຍັງຄົງຢູ່ໃນສ່ວນປາຍ. ມັນໄດ້ຖືກແນະນໍາໃຫ້ໃຊ້ຄໍາແນະນໍາທີ່ມີການດູດຊຶມຕໍ່າສໍາລັບການທົດລອງ.
4. ການເລືອກຕົວດັດແປງໃນການກໍ່ສ້າງຫ້ອງສະຸດ
ຊຸດການກໍ່ສ້າງຫ້ອງສະdoesຸດບໍ່ມີຕົວປະສົມນໍ້າຢາ, ແລະແນະ ນຳ ໃຫ້ໃຊ້ຊຸດນີ້ຮ່ວມກັບ TIANSeq ດັດຊະນີດັດຊະນີດ່ຽວ (Illumina) (4992641/4992642/4992378).
5. QC ຂອງຫ້ອງສະຸດ
ການກວດຫາປະລິມານໃນຫ້ອງສະຸດ: Qubit ແລະ qPCR ຖືກໃຊ້ເພື່ອ ກຳ ນົດຄວາມເຂັ້ມຂຸ້ນຂອງມະຫາຊົນແລະຄວາມເຂັ້ມຂຸ້ນຂອງ molar ຂອງຫ້ອງສະຸດຕາມ ລຳ ດັບ. ການດໍາເນີນງານແມ່ນສອດຄ່ອງກັບຄູ່ມືຜະລິດຕະພັນຢ່າງເຂັ້ມງວດ. ໂດຍທົ່ວໄປແລ້ວຄວາມເຂັ້ມຂົ້ນຂອງຫ້ອງສະຸດຈະຕອບສະ ໜອງ ໄດ້ຄວາມຮຽກຮ້ອງຕ້ອງການຂອງລໍາດັບ NGS. ການກວດຫາລະດັບການແຈກຢາຍຫ້ອງສະຸດ: ການໃຊ້ Agilent 2100 Bioanalyzer ເພື່ອກວດຫາຂອບເຂດການແຈກຢາຍຫ້ອງສະຸດ.
6. ການເລືອກcycleາຍເລກຮອບວຽນການຂະຫຍາຍ
ອີງຕາມຄໍາແນະນໍາ, ຈໍານວນຂອງຮອບວຽນ PCR ແມ່ນ 6-12, ແລະຈໍານວນຂອງຮອບວຽນ PCR ທີ່ຈໍາເປັນຄວນໄດ້ຮັບການຄັດເລືອກອີງຕາມການປ້ອນຕົວຢ່າງ. ຢູ່ໃນຫ້ອງສະຸດທີ່ໃຫ້ຜົນຜະລິດສູງ, ການຂະຫຍາຍຕົວຫຼາຍກວ່າປົກກະຕິເກີດຂື້ນໃນລະດັບແຕກຕ່າງກັນ, ເຊິ່ງສະແດງອອກໂດຍຈຸດສູງສຸດທີ່ໃຫຍ່ກວ່າເລັກນ້ອຍຫຼັງຈາກຈຸດສູງສຸດຂອງຊ່ວງເປົ້າinາຍໃນການກວດພົບ Agilent 2100 Bioanalyzer, ຫຼືຄວາມເຂັ້ມຂຸ້ນຂອງ Qubit ທີ່ກວດພົບຕໍ່າກວ່າ qPCR. ການຂະຫຍາຍສຽງເລັກນ້ອຍເປັນປະກົດການປົກກະຕິ, ເຊິ່ງບໍ່ມີຜົນຕໍ່ການຈັດລໍາດັບຫ້ອງສະandຸດແລະການວິເຄາະຂໍ້ມູນໃນພາຍຫຼັງ.
7. ຮວງປະກົດຢູ່ໃນໂປຣໄຟລ detection ການກວດພົບຂອງ Agilent 2100 Bioanalyzer
ລັກສະນະຂອງຮວງໃນ Agilent 2100 Bioanalyzer ກວດຫາແມ່ນເນື່ອງມາຈາກການແຍກຕົວຢ່າງທີ່ບໍ່ສະໍ່າສະເ,ີ, ບ່ອນທີ່ຈະມີຊິ້ນສ່ວນຫຼາຍຂຶ້ນຢູ່ໃນຂະ ໜາດ ທີ່ແນ່ນອນ, ແລະອັນນີ້ຈະກາຍເປັນທີ່ຈະແຈ້ງຫຼາຍຂຶ້ນຫຼັງຈາກການເສີມ PCR. ໃນກໍລະນີນີ້, ມັນຖືກແນະນໍາໃຫ້ບໍ່ດໍາເນີນການເລືອກຂະ ໜາດ, ieາຍຄວາມວ່າຕັ້ງເງື່ອນໄຂການແບ່ງສ່ວນໃຫ້ເປັນ 94 ° C ເປັນເວລາ 15 ນາທີ, ບ່ອນທີ່ການແຜ່ກະຈາຍຂອງຊິ້ນສ່ວນມີຂະ ໜາດ ນ້ອຍແລະເຂັ້ມຂຸ້ນ, ແລະສາມາດປັບປຸງຄວາມເປັນເອກະພາບກັນໄດ້.
ປະເພດຜະລິດຕະພັນ
ເປັນຫຍັງເລືອກພວກເຮົາ
ນັບຕັ້ງແຕ່ການສ້າງຕັ້ງ, ໂຮງງານຂອງພວກເຮົາໄດ້ພັດທະນາຜະລິດຕະພັນລະດັບໂລກອັນທໍາອິດໂດຍຍຶດັ້ນຫຼັກການ
ຂອງຄຸນນະພາບຄັ້ງທໍາອິດ. ຜະລິດຕະພັນຂອງພວກເຮົາໄດ້ຮັບຊື່ສຽງໂດ່ງດັງໃນອຸດສາຫະກໍາແລະເປັນທີ່ເຊື່ອຖືໄດ້ໃນບັນດາລູກຄ້າໃand່ແລະເກົ່າ.








